Arbeitsgruppe Professor Rademann
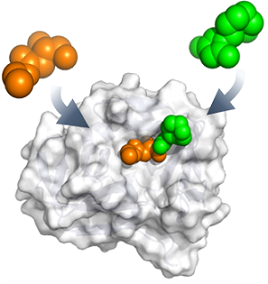
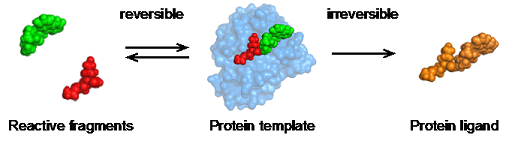
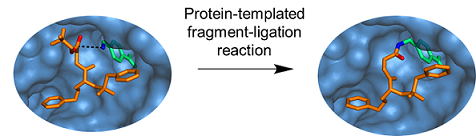
Wir, die Arbeitsgruppe Rademann, entwickeln Methoden für die Identifizierung und Optimierung von Protein-Liganden durch Fragment-Ligationsassays. In diesem Verfahren reagieren kleine Protein-bindende Moleküle katalysiert durch die Proteinoberfläche miteinander und bilden so stärker bindende Proteinliganden.
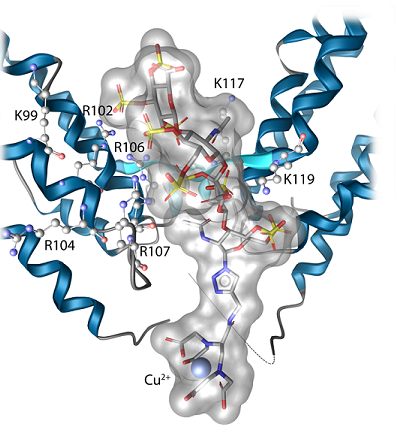
Das Verfahren detektiert die Proteinbindung von kleinen Molekülen mit besonderer Empfindlichkeit und an genau definierten Positionen der Proteinoberfläche. Ziel unser Arbeiten ist es, auf diese Weise besonders effiziente Wirkstoffmoleküle zu synthetisieren und zu entwickeln. Unsere Methoden erlauben uns im Detail zu verstehen, welche Bausteine eines Moleküls Beiträge zur Proteinbindung leisten und die Bindung dadurch kooperativ verstärken.
Die neuen Wirkstoffmoleküle werden anschließend für die strukturelle und funktionelle Charakterisierung von Proteinen, für die Visualisierung von molekularen Wechselwirkungen in lebenden Zellen und für die pharmakologische Validierung verwendet.
Die Arbeitsgruppe kombiniert dazu Methoden der chemischen Synthese, mit bioanalytischen, biochemischen und biophysikalischen Techniken.
Unsere Proteintargets umfassen pharmakologisch relevante Proteine bei den Infektionserkrankungen und Krebs, vor allem Proteasen, Phosphatasen, Protein-Protein und Protein-Kohlenhydrat-Interaktionen.